 Una revisión sobre los aspectos técnicos y utilidad de la tecnología denominada single-cell genome sequencing, parte de una serie sobre el tema, puede ser consultada en Gawad C, Koh W, Quake SR. Single-cell genome sequencing: current state of the science. Nature Reviews Genetics 2016; doi:10.1038/nrg.2015.16.
Una revisión sobre los aspectos técnicos y utilidad de la tecnología denominada single-cell genome sequencing, parte de una serie sobre el tema, puede ser consultada en Gawad C, Koh W, Quake SR. Single-cell genome sequencing: current state of the science. Nature Reviews Genetics 2016; doi:10.1038/nrg.2015.16.
 Web-based Gene Pathogenicity Analysis (WGPA) es una herramienta para determinar cuánto pueden aportar al riesgo de enfermar las mutaciones encontradas en los genomas individuales. Sus características y aplicaciones son descritas en Diaz-Montana JJ, Rackham OJ, Diaz-Diaz N, Petretto E. Web-based Gene Pathogenicity Analysis (WGPA): a web platform to interpret gene pathogenicity from personal genome data. Bioinformatics. 2016 Feb 15;32(4):635-7.
Web-based Gene Pathogenicity Analysis (WGPA) es una herramienta para determinar cuánto pueden aportar al riesgo de enfermar las mutaciones encontradas en los genomas individuales. Sus características y aplicaciones son descritas en Diaz-Montana JJ, Rackham OJ, Diaz-Diaz N, Petretto E. Web-based Gene Pathogenicity Analysis (WGPA): a web platform to interpret gene pathogenicity from personal genome data. Bioinformatics. 2016 Feb 15;32(4):635-7.
Filed under Algoritmos, Bioinformática, Polimorfismos by on . Comment. ![]()
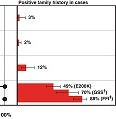 La comparación entre casos con enfermedad por priones y dos grupos de controles ha revelado una prevalencia poblacional de los genes patogénicos mucho mayor que la esperada. Revise el estudio en Minikel EV, Vallabh SM, Lek M, Estrada K, Samocha KE, Sathirapongsasuti JF, et al. Quantifying prion disease penetrance using large population control cohorts. Science Translational Medicine 2016;8(322):322ra9.
La comparación entre casos con enfermedad por priones y dos grupos de controles ha revelado una prevalencia poblacional de los genes patogénicos mucho mayor que la esperada. Revise el estudio en Minikel EV, Vallabh SM, Lek M, Estrada K, Samocha KE, Sathirapongsasuti JF, et al. Quantifying prion disease penetrance using large population control cohorts. Science Translational Medicine 2016;8(322):322ra9.
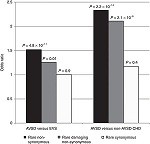 Tras la secuenciación del exoma, al menos seis genes (NIPBL, CHD7, CEP152, BMPR1a, ZFPM2 y MDM4) se asociaron con el defecto del septo atrioventricular, de acuerdo con D’Alessandro LCA, Al Turki S, Manickaraj AK, Manase D, Mulder BJM, Bergin L, et al. Exome sequencing identifies rare variants in multiple genes in atrioventricular septal defect. Genetics in Medicine 2016;18:189–198.
Tras la secuenciación del exoma, al menos seis genes (NIPBL, CHD7, CEP152, BMPR1a, ZFPM2 y MDM4) se asociaron con el defecto del septo atrioventricular, de acuerdo con D’Alessandro LCA, Al Turki S, Manickaraj AK, Manase D, Mulder BJM, Bergin L, et al. Exome sequencing identifies rare variants in multiple genes in atrioventricular septal defect. Genetics in Medicine 2016;18:189–198.
Filed under Cardiopatías, Defectos congénitos, Diagnóstico, Polimorfismos by on . Comment. ![]()
 A partir de los polimorfismos mononucleotídicos compartidos, se ha estimado un vínculo poligénico entre el reflujo gastroesofágico y el adenocarcinoma de ese órgano. Tal es la conclusión en Gharahkhani P, Tung J, Hinds D, Mishra A, Barrett’s and Esophageal Adenocarcinoma Consortium (BEACON), Vaughan TL, et al. Chronic gastroesophageal reflux disease shares genetic background with esophageal adenocarcinoma and Barrett’s esophagus. Hum. Mol. Genet. 2016;25(4):828-835.
A partir de los polimorfismos mononucleotídicos compartidos, se ha estimado un vínculo poligénico entre el reflujo gastroesofágico y el adenocarcinoma de ese órgano. Tal es la conclusión en Gharahkhani P, Tung J, Hinds D, Mishra A, Barrett’s and Esophageal Adenocarcinoma Consortium (BEACON), Vaughan TL, et al. Chronic gastroesophageal reflux disease shares genetic background with esophageal adenocarcinoma and Barrett’s esophagus. Hum. Mol. Genet. 2016;25(4):828-835.
Filed under Cáncer, Polimorfismos by on . Comment. ![]()
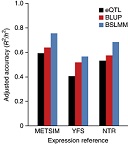 Una estrategia que integra las mediciones de expresión génica con los estudios de asociación condujo a la identificación de 69 genes relacionados con la obesidad. Consulte el trabajo en Gusev A, Ko A, Shi H, Bhatia G, Chung W, Penninx BWJH, et al. Integrative approaches for large-scale transcriptome-wide association studies. Nature Genetics 2016; doi:10.1038/ng.3506.
Una estrategia que integra las mediciones de expresión génica con los estudios de asociación condujo a la identificación de 69 genes relacionados con la obesidad. Consulte el trabajo en Gusev A, Ko A, Shi H, Bhatia G, Chung W, Penninx BWJH, et al. Integrative approaches for large-scale transcriptome-wide association studies. Nature Genetics 2016; doi:10.1038/ng.3506.
Filed under Diagnóstico, Polimorfismos by on . Comment. ![]()
– Yin H, Song CQ, Dorkin JR, Zhu LJ, Li Y, Wu Q, et al. Therapeutic genome editing by combined viral and non-viral delivery of CRISPR system components in vivo. Nature Biotechnology 2016; doi:10.1038/nbt.3471.
– Yang Y, Wang L, Bell P, McMenamin D, He Z, White J, et al. A dual AAV system enables the Cas9-mediated correction of a metabolic liver disease in newborn mice. Nature Biotechnology 2016; doi:10.1038/nbt.3469.
– Lee CM, Cradick TJ, Bao G. The Neisseria meningitidis CRISPR-Cas9 System Enables Specific Genome Editing in Mammalian Cells. Molecular Therapy 2016; doi: 10.1038/mt.2016.8.
Filed under Genómica, Patógenos, Publicaciones, Tratamientos by on . Comment. ![]()
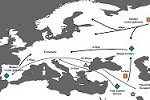 Los hallazgos en torno a la secuenciación del ADN antiguo han aportado nuevas evidencias de las migraciones y las mezclas que han dado forma a las poblaciones humanas. El tema es revisado en Haber M, Mezzavilla M, Xue Y, Tyler-Smith C. Ancient DNA and the rewriting of human history: be sparing with Occam’s razor. Genome Biology 2016;17:1.
Los hallazgos en torno a la secuenciación del ADN antiguo han aportado nuevas evidencias de las migraciones y las mezclas que han dado forma a las poblaciones humanas. El tema es revisado en Haber M, Mezzavilla M, Xue Y, Tyler-Smith C. Ancient DNA and the rewriting of human history: be sparing with Occam’s razor. Genome Biology 2016;17:1.
Filed under Filogenética, Genómica, Polimorfismos by on . Comment. ![]()
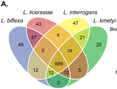 El análisis genómico de 20 especies de Leptospira ha encontrado algunas características que hacen a la bacteria patogénica, como las proteínas modificadoras de la virulencia y el sistema CRISPR/Cas. Los detalles pueden leerse en Fouts DE, Matthias MA, Adhikarla H, Adler B, Amorim-Santos L, Berg DE, et al. What Makes a Bacterial Species Pathogenic?: Comparative Genomic Analysis of the Genus Leptospira. PLoS Negl Trop Dis 2016;10(2):e0004403.
El análisis genómico de 20 especies de Leptospira ha encontrado algunas características que hacen a la bacteria patogénica, como las proteínas modificadoras de la virulencia y el sistema CRISPR/Cas. Los detalles pueden leerse en Fouts DE, Matthias MA, Adhikarla H, Adler B, Amorim-Santos L, Berg DE, et al. What Makes a Bacterial Species Pathogenic?: Comparative Genomic Analysis of the Genus Leptospira. PLoS Negl Trop Dis 2016;10(2):e0004403.
Filed under Diagnóstico, Filogenética, Patógenos, Polimorfismos by on . Comment. ![]()
 Tres genes (GBP5, DUSP3 y KLF2) pueden ser la clave para diagnosticar con una prueba de sangre total la tuberculosis pulmonar activa, según concluyen los autores del artículo Sweeney TE, Braviak L, Tato CM, Khatri P. Genome-wide expression for diagnosis of pulmonary tuberculosis: a multicohort analysis. The Lancet Respiratory Medicine 19 February 2016; DOI: http://dx.doi.org/10.1016/S2213-2600(16)00048-5.
Tres genes (GBP5, DUSP3 y KLF2) pueden ser la clave para diagnosticar con una prueba de sangre total la tuberculosis pulmonar activa, según concluyen los autores del artículo Sweeney TE, Braviak L, Tato CM, Khatri P. Genome-wide expression for diagnosis of pulmonary tuberculosis: a multicohort analysis. The Lancet Respiratory Medicine 19 February 2016; DOI: http://dx.doi.org/10.1016/S2213-2600(16)00048-5.
Filed under Diagnóstico, Patógenos by on . Comment. ![]()


