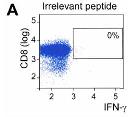
 La combinación de secuenciación del exoma y metodologías bioinformáticas condujo a predecir antígenos de neoformación en las leucemias, verificar su unión experimental a HLA y detectar linfocitos T CD8+ específicos en los pacientes afectados. Lea sobre estas tecnologías y sus resultados en Rajasagi M, Shukla SA, Fritsch EF, Keskin DB, DeLuca D, Carmona E, et al. Systematic identification of personal tumor-specific neoantigens in chronic lymphocytic leukemia. Blood. 2014 Jun 2. pii: blood-2014-04-567933.
La combinación de secuenciación del exoma y metodologías bioinformáticas condujo a predecir antígenos de neoformación en las leucemias, verificar su unión experimental a HLA y detectar linfocitos T CD8+ específicos en los pacientes afectados. Lea sobre estas tecnologías y sus resultados en Rajasagi M, Shukla SA, Fritsch EF, Keskin DB, DeLuca D, Carmona E, et al. Systematic identification of personal tumor-specific neoantigens in chronic lymphocytic leukemia. Blood. 2014 Jun 2. pii: blood-2014-04-567933.
Filed under Cáncer, Diagnóstico, Inmunoinformática by on . Comment. ![]()





