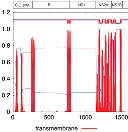
 Las epidemias de peste bubónica se prolongaron en Europa al menos tres siglos más después de la pandemia de muerte negra, de acuerdo con secuencias genómicas de Yersinia pestis del siglo XVIII. El estudio puede ser consultado en Bos KI, Herbig A, Sahl J, Waglechner N, Fourment M, Forrest SA, et al. Eighteenth century Yersinia pestis genomes reveal the long-term persistence of an historical plague focus. eLife 2016;5:e12994.
Las epidemias de peste bubónica se prolongaron en Europa al menos tres siglos más después de la pandemia de muerte negra, de acuerdo con secuencias genómicas de Yersinia pestis del siglo XVIII. El estudio puede ser consultado en Bos KI, Herbig A, Sahl J, Waglechner N, Fourment M, Forrest SA, et al. Eighteenth century Yersinia pestis genomes reveal the long-term persistence of an historical plague focus. eLife 2016;5:e12994.
Filed under Bioinformática, Filogenética, Patógenos by on . Comment. ![]()