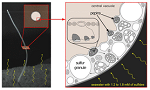
Un virus gigante envuelto encontrado en una ciénaga brasileña, ha sido llamado naiavirus y tiene un genoma de casi un millón de pares de bases. Su peculiar morfología destaca por tener una envoltura que se extiende cual cola, según se aprecia en Rodrigues M, Queiroz V, Arantes T, Limborço H, Neiva B, Arias N, et al. Naiavirus: an enveloped giant virus with a pleomorphic, flexible tail. Nature Communications, 2025;16:8306.
Un virus gigante envuelto encontrado en una ciénaga brasileña, ha sido llamado naiavirus y tiene un genoma de casi un millón de pares de bases. Su peculiar morfología destaca por tener una envoltura que se extiende cual cola, según se aprecia en Rodrigues M, Queiroz V, Arantes T, Limborço H, Neiva B, Arias N, et al. Naiavirus: an enveloped giant virus with a pleomorphic, flexible tail. Nature Communications, 2025;16:8306.
Filed under Genómica, Otros Proyectos Genoma by on . Comment. ![]()