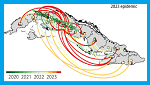
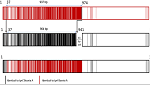
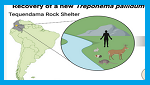
 En los restos de un individuo fallecido hace unos 5500 años en un sitio correspondiente a la actual Colombia, fue identificado el ADN de Treponema pallidum, en ausencia de signos de sífilis. Los resultados de los análisis genómicos y filogenéticos son descritos en Bozzi D, Broomandkhoshbacht NZ, Delgado M, Buikstra JE, Amorim CEG, Kassadjikova K, et al. A 5500-year-old Treponema pallidum genome from Sabana de Bogotá, Colombia. Science, 2026;391(6783).
En los restos de un individuo fallecido hace unos 5500 años en un sitio correspondiente a la actual Colombia, fue identificado el ADN de Treponema pallidum, en ausencia de signos de sífilis. Los resultados de los análisis genómicos y filogenéticos son descritos en Bozzi D, Broomandkhoshbacht NZ, Delgado M, Buikstra JE, Amorim CEG, Kassadjikova K, et al. A 5500-year-old Treponema pallidum genome from Sabana de Bogotá, Colombia. Science, 2026;391(6783).
Filed under Diagnóstico, Filogenética, Patógenos by on . Comment. ![]()