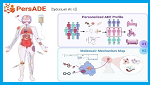
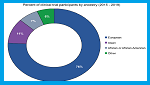
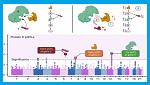
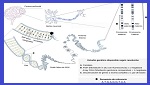
84 nuevas bases de datos y 86 actualizaciones son presentadas en el número Database de enero 2026, de la revista Nucleic Acids Research, que cubre genes, transcriptomas, enzimas, otras proteínas y haplotipos. Puede acceder a la descripción de esta edición en Rigden DJ, Fernández XM. The 2026 Nucleic Acids Research database issue and the online molecular biology database collection. Nucleic Acids Research, 2025;gkaf1427, https://doi.org/10.1093/nar/gkaf1427
84 nuevas bases de datos y 86 actualizaciones son presentadas en el número Database de enero 2026, de la revista Nucleic Acids Research, que cubre genes, transcriptomas, enzimas, otras proteínas y haplotipos. Puede acceder a la descripción de esta edición en Rigden DJ, Fernández XM. The 2026 Nucleic Acids Research database issue and the online molecular biology database collection. Nucleic Acids Research, 2025;gkaf1427, https://doi.org/10.1093/nar/gkaf1427
Filed under Bases de datos, Publicaciones by on . Comment. ![]()