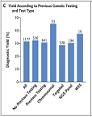
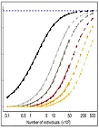
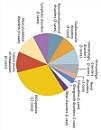
Una encuesta entre 238 expertos en enfermedades raras, reveló que el 87,9 % respaldó que el uso de la secuenciación neonatal debe estar disponible para todos los casos sospechosos de enfermedad monogénica tratable. Tal es el reporte en Gold NB, Adelson SM, Shah N, Williams S, Bick SL, Zoltick ES, et al. Perspectives of Rare Disease Experts on Newborn Genome Sequencing. JAMA Netw Open. 2023;6(5):e2312231.
Una encuesta entre 238 expertos en enfermedades raras, reveló que el 87,9 % respaldó que el uso de la secuenciación neonatal debe estar disponible para todos los casos sospechosos de enfermedad monogénica tratable. Tal es el reporte en Gold NB, Adelson SM, Shah N, Williams S, Bick SL, Zoltick ES, et al. Perspectives of Rare Disease Experts on Newborn Genome Sequencing. JAMA Netw Open. 2023;6(5):e2312231.
Filed under Defectos congénitos, Diagnóstico, Genómica, Medicina personalizada by on . Comment. ![]()