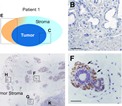
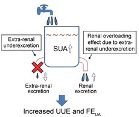
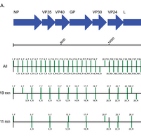
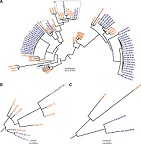
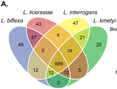
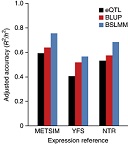
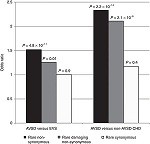
Numerosas variantes genéticas ubicadas en regiones reguladoras de la región HLA clase II incrementan la expresión de moléculas vinculadas a la actividad en el lupus eritematoso sistémico. Así puede leerse en Raj P, Rai E, Song R, Khan S, Wakeland BE, Viswanathan K, Carlos Arana, et al. Regulatory polymorphisms modulate the expression of HLA class II molecules and promote autoimmunity. eLife 2016;5:e12089.
Numerosas variantes genéticas ubicadas en regiones reguladoras de la región HLA clase II incrementan la expresión de moléculas vinculadas a la actividad en el lupus eritematoso sistémico. Así puede leerse en Raj P, Rai E, Song R, Khan S, Wakeland BE, Viswanathan K, Carlos Arana, et al. Regulatory polymorphisms modulate the expression of HLA class II molecules and promote autoimmunity. eLife 2016;5:e12089.
Filed under Diagnóstico, Genómica, Polimorfismos by on . Comment. ![]()