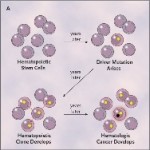
Los genes KIR2DS5 y KIR2DL1, ubicados en la región MHC del cromosoma 6, protegen de la pre-eclampsia a las mujeres de poblaciones subsaharianas. Secuencias diferentes reducen el riesgo en gestantes europeas, mientras que el genotipo KIR AA es un factor de riesgo, de acuerdo con Nakimulia A, Chazara O, Hiby SE, Farrell L, Tukwasibwe S, Jayaraman J, et al. A KIR B centromeric region present in Africans but not Europeans protects pregnant women from pre-eclampsia. PNAS January 5, 2015; doi: 10.1073/pnas.1413453112.
Los genes KIR2DS5 y KIR2DL1, ubicados en la región MHC del cromosoma 6, protegen de la pre-eclampsia a las mujeres de poblaciones subsaharianas. Secuencias diferentes reducen el riesgo en gestantes europeas, mientras que el genotipo KIR AA es un factor de riesgo, de acuerdo con Nakimulia A, Chazara O, Hiby SE, Farrell L, Tukwasibwe S, Jayaraman J, et al. A KIR B centromeric region present in Africans but not Europeans protects pregnant women from pre-eclampsia. PNAS January 5, 2015; doi: 10.1073/pnas.1413453112.
Filed under Diagnóstico, Polimorfismos by on . Comment. ![]()