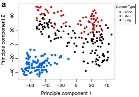
La secuenciación del exoma en 13 trios de padres y su descendencia, así como de 112 controles, condujo a la identificación de cinco variantes del factor de transcripción NR2F2, asociados a defectos no sindrómicos del septo auriculoventricular . Puede acceder al reporte completo en Al Turki S, Manickaraj AK, Mercer CL, Gerety SS, Hitz MP, Lindsay S, et al. Rare Variants in NR2F2 Cause Congenital Heart Defects in Humans. The American Journal of Human Genetics 3 April 2014;94(4):574–585.
La secuenciación del exoma en 13 trios de padres y su descendencia, así como de 112 controles, condujo a la identificación de cinco variantes del factor de transcripción NR2F2, asociados a defectos no sindrómicos del septo auriculoventricular . Puede acceder al reporte completo en Al Turki S, Manickaraj AK, Mercer CL, Gerety SS, Hitz MP, Lindsay S, et al. Rare Variants in NR2F2 Cause Congenital Heart Defects in Humans. The American Journal of Human Genetics 3 April 2014;94(4):574–585.
Filed under Cardiopatías, Defectos congénitos, Diagnóstico, Polimorfismos by on . Comment. ![]()