La combinación de técnicas genómicas y bioinformáticas en sujetos con diferente ascendencia, en este caso con ancestros europeos o asiáticos, reveló dos nuevas variantes (rs10816625 y rs13294895) y sus efectos en la génesis del cáncer de mama. El resumen del hallazgo está disponible en Orr N, Dudbridge F, Dryden N, Maguire S, Novo D, Perrakis E, et al. Fine-mapping identifies two additional breast cancer susceptibility loci at 9q31.2. Hum. Mol. Genet. 2015; doi: 10.1093/hmg/ddv035.
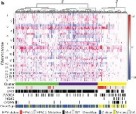 Los genes y vías implicados en los carcinomas escamosos de cabeza y cuello difieren entre los tumores relacionados con papilomavirus o el hábito de fumar. Sucede algo similar según la localización anatómica, sea en la cavidad bucal o la laringe, según se lee en The Cancer Genome Atlas Network. Comprehensive genomic characterization of head and neck squamous cell carcinomas. Nature 2015;517:576–582.
Los genes y vías implicados en los carcinomas escamosos de cabeza y cuello difieren entre los tumores relacionados con papilomavirus o el hábito de fumar. Sucede algo similar según la localización anatómica, sea en la cavidad bucal o la laringe, según se lee en The Cancer Genome Atlas Network. Comprehensive genomic characterization of head and neck squamous cell carcinomas. Nature 2015;517:576–582.
Filed under Cáncer, Diagnóstico, Genómica by on . Comment. ![]()
 La Agencia de Medicamentos y Alimentos (FDA por sus siglas en inglés) de Estados Unidos realizará en el próximo mes de febrero un taller para discutir los aspectos regulatorios de los ensayos diagnósticos basados en la secuenciación de próxima generación (NGS en inglés). El documento marco para el debate está ya disponible.
La Agencia de Medicamentos y Alimentos (FDA por sus siglas en inglés) de Estados Unidos realizará en el próximo mes de febrero un taller para discutir los aspectos regulatorios de los ensayos diagnósticos basados en la secuenciación de próxima generación (NGS en inglés). El documento marco para el debate está ya disponible.
Filed under Diagnóstico, Eventos, Genómica by on . Comment. ![]()
 Al tiempo que se requiere estudiar las dimensiones clínicas, tecnológicas, reguladoras y bioéticas de la medicina genómica, resulta imprescindible la preparación inmediata del personal sanitario para la introducción y la extensión de estas tecnologías en el ejercicio de su práctica profesional. Así se comenta en Hernández-Betancourt JC, Serrano-Barrera OR. La medicina personalizada, la revolución genómica y el Sistema Nacional de Salud. Rev Cubana Salud Pública. 2014;40(4).
Al tiempo que se requiere estudiar las dimensiones clínicas, tecnológicas, reguladoras y bioéticas de la medicina genómica, resulta imprescindible la preparación inmediata del personal sanitario para la introducción y la extensión de estas tecnologías en el ejercicio de su práctica profesional. Así se comenta en Hernández-Betancourt JC, Serrano-Barrera OR. La medicina personalizada, la revolución genómica y el Sistema Nacional de Salud. Rev Cubana Salud Pública. 2014;40(4).
Filed under Bioética, Capacitación, Diagnóstico, Genómica, Publicaciones by on . Comment. ![]()
El ADN genómico de una mola hidatiforme haploide ha sido secuenciado y su ensamblaje reportado a la base de datos AssemblyDB. Los detalles y objetivos son declarados en Steinberg KM, Schneider VA, Graves-Lindsay TA, Fulton RS, Agarwala R, Huddleston J, et al. Single haplotype assembly of the human genome from a hydatidiform mole. Genome Res. November 4, 2014; doi: 10.1101/gr.180893.114.
Filed under Diagnóstico, Genómica by on . Comment. ![]()
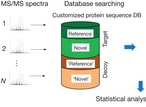 La integración de las técnicas genómicas y proteómicas, que incluye la generación de bases de datos, incrementa las posibilidades de identificación de proteínas y la definición de modelos de interacción entre genes. El tema puede ser revisado en Nesvizhskii AI. Proteogenomics: concepts, applications and computational strategies. Nature Methods 2014;11:1114–1125.
La integración de las técnicas genómicas y proteómicas, que incluye la generación de bases de datos, incrementa las posibilidades de identificación de proteínas y la definición de modelos de interacción entre genes. El tema puede ser revisado en Nesvizhskii AI. Proteogenomics: concepts, applications and computational strategies. Nature Methods 2014;11:1114–1125.
Filed under Diagnóstico, Genómica, Proteómica by on . Comment. ![]()
 Los kits para la extracción de ADN y otros reactivos son una fuente de contaminación frecuente en los estudios de secuenciación del microbioma. Algunas recomendaciones para mitigar los efectos indeseables de este fenómeno son propuestas en Salter SJ, Cox MJ, Turek EM, Calus ST, Cookson WO, Moffatt MF, et al. Reagent and laboratory contamination can critically impact sequence-based microbiome analyses. BMC Biology 2014;12:87.
Los kits para la extracción de ADN y otros reactivos son una fuente de contaminación frecuente en los estudios de secuenciación del microbioma. Algunas recomendaciones para mitigar los efectos indeseables de este fenómeno son propuestas en Salter SJ, Cox MJ, Turek EM, Calus ST, Cookson WO, Moffatt MF, et al. Reagent and laboratory contamination can critically impact sequence-based microbiome analyses. BMC Biology 2014;12:87.
Filed under Diagnóstico, Genómica, Microbioma by on . Comment. ![]()
 La inclusión de un parámetro adicional en un nuevo modelo que incrementa la capacidad para definir la asociación en los estudios de genoma completo, selecciona la mejor combinación entre los algoritmos de parentesco y agrupación. La nueva propuesta es descrita en Li M, Liu X, Bradbury P, Yu J, Zhang Y-M, Todhunter RJ, et al. Enrichment of statistical power for genome-wide association studies. BMC Biology 2014;12:73.
La inclusión de un parámetro adicional en un nuevo modelo que incrementa la capacidad para definir la asociación en los estudios de genoma completo, selecciona la mejor combinación entre los algoritmos de parentesco y agrupación. La nueva propuesta es descrita en Li M, Liu X, Bradbury P, Yu J, Zhang Y-M, Todhunter RJ, et al. Enrichment of statistical power for genome-wide association studies. BMC Biology 2014;12:73.
Filed under Algoritmos, Bioinformática, Diagnóstico, Genómica by on . Comment. ![]()
 La revista Nature Reviews Genetics ha publicado una selección de artículos sobre las aplicaciones de la secuenciación de próxima generación (del inglés next–generation sequencing) y el impacto que ha tenido en el análisis genómico y el estudio de las interacciones entre proteínas y ácidos nucleicos.
La revista Nature Reviews Genetics ha publicado una selección de artículos sobre las aplicaciones de la secuenciación de próxima generación (del inglés next–generation sequencing) y el impacto que ha tenido en el análisis genómico y el estudio de las interacciones entre proteínas y ácidos nucleicos.
Filed under Diagnóstico, Genómica by on . Comment. ![]()
![]() Las escuelas de Medicina e Ingeniería de la Universidad de Stanford han puesto en práctica un programa profesional certificado en línea sobre genética y genómica. Se incluyen cursos electivos sobre biotecnología, células madre, cáncer, terapia génica y farmacogenómica. Los contenidos y procedimientos están disponibles en Stanford Genetics and Genomics Certificate.
Las escuelas de Medicina e Ingeniería de la Universidad de Stanford han puesto en práctica un programa profesional certificado en línea sobre genética y genómica. Se incluyen cursos electivos sobre biotecnología, células madre, cáncer, terapia génica y farmacogenómica. Los contenidos y procedimientos están disponibles en Stanford Genetics and Genomics Certificate.
Filed under Capacitación, Genómica by on . Comment. ![]()



