 El análisis de las secuencias genómicas de muestras de virus Ébola arroja luz sobre los orígenes, la evolución y la diseminación del agente durante el brote 2013-2016 en África. Lea los detalles en Holmes EC, Dudas G, Rambaut A, Andersen KG. The evolution of Ebola virus: Insights from the 2013–2016 epidemic. Nature 2016;538:193–200.
El análisis de las secuencias genómicas de muestras de virus Ébola arroja luz sobre los orígenes, la evolución y la diseminación del agente durante el brote 2013-2016 en África. Lea los detalles en Holmes EC, Dudas G, Rambaut A, Andersen KG. The evolution of Ebola virus: Insights from the 2013–2016 epidemic. Nature 2016;538:193–200.
Filed under Bioinformática, Filogenética, Patógenos by on . Comment. ![]()
 Las evidencias sobre la diversidad fenotípica y los mecanismos genéticos subyacentes en la bacteria Neisseria meningitidis han sido reforzadas a partir de la secuenciación y comparación genómicas de colonias obtenidas en condiciones poco selectivas. Los hallazgos aparecen en Siena E, D’Aurizio R, Riley D, Tettelin H, Guidotti S, Torricelli G, et al. In-silico prediction and deep-DNA sequencing validation indicate phase variation in 115 Neisseria meningitidis genes. BMC Genomics 2016;17:843.
Las evidencias sobre la diversidad fenotípica y los mecanismos genéticos subyacentes en la bacteria Neisseria meningitidis han sido reforzadas a partir de la secuenciación y comparación genómicas de colonias obtenidas en condiciones poco selectivas. Los hallazgos aparecen en Siena E, D’Aurizio R, Riley D, Tettelin H, Guidotti S, Torricelli G, et al. In-silico prediction and deep-DNA sequencing validation indicate phase variation in 115 Neisseria meningitidis genes. BMC Genomics 2016;17:843.
Filed under Filogenética, Patógenos by on . Comment. ![]()
 Las variaciones en la expresión génica en 19 regiones cerebrales han aclarado que los cambios moleculares en la enfermedad de Alzheimer comienzan tempranamente en la afección y ubican a los giros del lóbulo temporal con las mayores y más precoces alteraciones. Así se publica en Wang M, Roussos P, McKenzie A, Zhou X, Kajiwara Y, Brennand KJ, et al. Integrative network analysis of nineteen brain regions identifies molecular signatures and networks underlying selective regional vulnerability to Alzheimer’s disease. Genome Medicine 2016;8:104.
Las variaciones en la expresión génica en 19 regiones cerebrales han aclarado que los cambios moleculares en la enfermedad de Alzheimer comienzan tempranamente en la afección y ubican a los giros del lóbulo temporal con las mayores y más precoces alteraciones. Así se publica en Wang M, Roussos P, McKenzie A, Zhou X, Kajiwara Y, Brennand KJ, et al. Integrative network analysis of nineteen brain regions identifies molecular signatures and networks underlying selective regional vulnerability to Alzheimer’s disease. Genome Medicine 2016;8:104.
Filed under Neurología, Polimorfismos by on . Comment. ![]()
– MIPEP recessive variants cause a syndrome of left ventricular non-compaction, hypotonia, and infantile death
– Genome-wide associations for birth weight and correlations with adult disease
– Identification of genomic loci associated with resting heart rate and shared genetic predictors with all-cause mortality
– Genome-wide analysis identifies 12 loci influencing human reproductive behavior
– Stabilizing mutations of KLHL24 ubiquitin ligase cause loss of keratin 14 and human skin fragility
Filed under Cardiopatías, Defectos congénitos, Diagnóstico, Polimorfismos by on . Comment. ![]()
A partir de muestras colectadas en más de 700 hogares en todo Estados Unidos, se ha abordado por medio de la secuenciación la diversidad y los potenciales riesgos para la salud humana de los artrópodos que habitan en las casas. Así se presenta en Madden AA, Barberán A, Bertone MA, Menninger HL, Dunn RR, Fierer N, et al. The diversity of arthropods in homes across the United States as determined by environmental DNA analyses. Molecular Ecology2016;25(21).
 Una revisión sobre las aplicaciones clínicas de la secuenciación de nueva generación, particularmente en la oncología y la medicina de precisión, así como sus potencialidades para mejorar el diagnóstico, el pronóstico y el tratamiento de pacientes oncológicos, está disponible en Calabria I, Pedrola L, Berlanga P, Aparisi MJ, Sánchez-Izquierdo D, Cañete A, et al. El nuevo reto en oncología: la secuenciación NGS y su aplicación a la medicina de precisión. An Pediatr 2016;85(5):273.
Una revisión sobre las aplicaciones clínicas de la secuenciación de nueva generación, particularmente en la oncología y la medicina de precisión, así como sus potencialidades para mejorar el diagnóstico, el pronóstico y el tratamiento de pacientes oncológicos, está disponible en Calabria I, Pedrola L, Berlanga P, Aparisi MJ, Sánchez-Izquierdo D, Cañete A, et al. El nuevo reto en oncología: la secuenciación NGS y su aplicación a la medicina de precisión. An Pediatr 2016;85(5):273.
Filed under Cáncer, Diagnóstico, Genómica, Medicina personalizada by on . Comment. ![]()
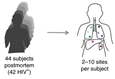 Las peculiaridades de la migración del Mycobacterium tuberculosis entre órganos de individuos con vih son divulgadas en Lieberman TD, Wilson D, Misra R, Xiong LL, Moodley P, Cohen T, et al. Genomic diversity in autopsy samples reveals within-host dissemination of HIV-associated Mycobacterium tuberculosis. Nature Medicine 2016; doi:10.1038/nm.4205. Mientras, la distribución global de una cepa de la bacteria es dilucidada en Stucki D, Brites D, Jeljeli L, Coscolla M, Liu Q, Trauner A, et al. Mycobacterium tuberculosis lineage 4 comprises globally distributed and geographically restricted sublineages. Nature Genetics 2016; doi:10.1038/ng.3704.
Las peculiaridades de la migración del Mycobacterium tuberculosis entre órganos de individuos con vih son divulgadas en Lieberman TD, Wilson D, Misra R, Xiong LL, Moodley P, Cohen T, et al. Genomic diversity in autopsy samples reveals within-host dissemination of HIV-associated Mycobacterium tuberculosis. Nature Medicine 2016; doi:10.1038/nm.4205. Mientras, la distribución global de una cepa de la bacteria es dilucidada en Stucki D, Brites D, Jeljeli L, Coscolla M, Liu Q, Trauner A, et al. Mycobacterium tuberculosis lineage 4 comprises globally distributed and geographically restricted sublineages. Nature Genetics 2016; doi:10.1038/ng.3704.
Filed under Filogenética, Patógenos by on . Comment. ![]()
 El estudio de las células tumorales circulantes en pacientes con mieloma múltiple puede aportar la misma o mayor información que las muestras de médula ósea, incluida la clasificación y el pronóstico de la enfermedad. El resumen del artículo está disponible en Lohr JG, Kim S, Gould J, Knoechel B, Drier Y, Cotton MJ, et al. Genetic interrogation of circulating multiple myeloma cells at single-cell resolution. Science Translational Medicine 02 Nov 2016;8(363):363ra147.
El estudio de las células tumorales circulantes en pacientes con mieloma múltiple puede aportar la misma o mayor información que las muestras de médula ósea, incluida la clasificación y el pronóstico de la enfermedad. El resumen del artículo está disponible en Lohr JG, Kim S, Gould J, Knoechel B, Drier Y, Cotton MJ, et al. Genetic interrogation of circulating multiple myeloma cells at single-cell resolution. Science Translational Medicine 02 Nov 2016;8(363):363ra147.
Filed under Cáncer, Diagnóstico, Genómica by on . Comment. ![]()
Un proceder de secuenciación de segunda generación para ADN plasmático identificó una mutación de novo en BRAF en un feto con síndrome cardiofaciocutáneo, según se reporta en Chan KCA, Jiang P, Sun K, Cheng YKY, Tong YK, Chen SH, et al. Second generation noninvasive fetal genome analysis reveals de novo mutations, single-base parental inheritance, and preferred DNA ends. PNAS 2016; doi: 10.1073/pnas.1615800113. Otro estudio revela la posibilidad de análisis del genoma fetal a partir de células trofoblásticas en muestras obtenidas por citología vaginal, en una gestación de apenas cinco semanas: Jain CV, Kadam L, van Dijk M, Kohan-Ghadr HR, Kilburn BA, Hartman C, et al. Fetal genome profiling at 5 weeks of gestation after noninvasive isolation of trophoblast cells from the endocervical canal. Science Translational Medicine 2016;8(363):363re4.
Filed under Defectos congénitos, Diagnóstico by on . Comment. ![]()


