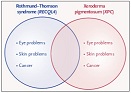
Un resumen comentado sobre los artículos más leídos y discutidos durante el año que termina, con temas sobre genómica, epigenómica, tecnologías y enfermedades humanas, es presentado por la revista Genome Biology en su Best of 2016: Top Picks from Genome Biology.
Un resumen comentado sobre los artículos más leídos y discutidos durante el año que termina, con temas sobre genómica, epigenómica, tecnologías y enfermedades humanas, es presentado por la revista Genome Biology en su Best of 2016: Top Picks from Genome Biology.
Filed under Publicaciones by on . Comment. ![]()