Las principales fuentes de variabilidad en el análisis de datos de varias tecnologías ómicas por medio de máquinas de soporte vectorial es tema del artículo Han H. Diagnostic biases in translational bioinformatics. BMC Medical Genomics 2015;8:46.
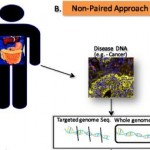 La presencia en el genoma de un individuo, cuando se le toma como propia referencia, de una variación en células somáticas o también en línea germinal es crucial para definirla como mutación. Así se fundamenta en Karki R, Pandya D, Elston RC, Ferlini C. Defining “mutation” and “polymorphism” in the era of personal genomics. BMC Medical Genomics 2015;8:37.
La presencia en el genoma de un individuo, cuando se le toma como propia referencia, de una variación en células somáticas o también en línea germinal es crucial para definirla como mutación. Así se fundamenta en Karki R, Pandya D, Elston RC, Ferlini C. Defining “mutation” and “polymorphism” in the era of personal genomics. BMC Medical Genomics 2015;8:37.
Filed under Bioinformática, Diagnóstico, Polimorfismos by on . Comment. ![]()
Las técnicas de análisis de las redes de genes y vías de señalización implicadas en la génesis y progresión de los tumores malignos son comentadas en Creixell P, Reimand J, Haider S, Wu G, Shibata T, Vazquez M, et al. Pathway and network analysis of cancer genomes. Nature Methods 2015;12:615–621.
Filed under Bioinformática, Biología de sistemas, Cáncer, Diagnóstico by on . Comment. ![]()
Además de un mapa de la tasa de mutaciones de novo en todo el genoma, un reciente estudio muestra, entre otras características, que los cambios son más numerosos y frecuentes en la descendencia de padres de mayor edad. Lea otros resultados en Francioli LC, Polak PP, Koren A, Menelaou A, Chun S, Renkens I, et al. Genome-wide patterns and properties of de novo mutations in humans. Nature Genetics 2015;47:822–826.
Filed under Bioinformática, Diagnóstico, Genómica, Polimorfismos by on . Comment. ![]()
Por medio de la espectrometría de masa se ha revelado una red de 23744 interacciones entre 7668 proteínas en una línea celular, la mayoría de ellas no documentadas hasta ahora. Algunas aplicaciones médicas de este resultado son comentadas en Huttlin EL, Ting L, Bruckner RJ, Gebreab F, Gygi MP, Szpyt J, et al. The BioPlex Network: A Systematic Exploration of the Human Interactome. Cell 16 July 2015;162(2):425–440.
Filed under Bioinformática, Biología de sistemas, Proteómica by on . Comment. ![]()
EpiFactors es una base de datos que contiene información sobre 815 proteínas con funciones en la regulación epigenética de la expresión de genes y la estabilidad del genoma. Sus características y contenidos son abordados en Medvedeva YA, Lennartsson A, Ehsani R, Kulakovskiy IV, Vorontsov IE, Panahandeh P, et al. EpiFactors: a comprehensive database of human epigenetic factors and complexes. Database (Oxford). 2015 Jul 7;2015. doi: 10.1093/database/bav067.
Filed under Bases de datos, Bioinformática, Epigenoma by on . Comment. ![]()
El catálogo de estudios de asociación de genoma completo (genome-wide association studies, GWAS) ha pasado a ser hospedado por el Instituto Europeo de Bioinformática. La colección de asociaciones entre secuencias de ADN y rasgos humanos ha sido rediseñado para facilitar la búsqueda por profesionales de la biología y la medicina, y puede consultarse ahora aquí.
Filed under Bases de datos, Bioinformática, Genómica by on . Comment. ![]()
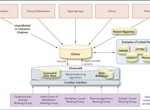 La información que se acumula y actualiza en las bases de datos con resultados de los estudios genómicos es un recurso valioso que puede complementar el diagnóstico médico y orientar en muchos casos la conducta a seguir. Así se ejemplifica y argumenta en Rehm HL, Berg JS, Brooks LD, Bustamante CD, Evans JP, Landrum MJ, et al. ClinGen — The Clinical Genome Resource. N Engl J Med 2015;372:2235-2242.
La información que se acumula y actualiza en las bases de datos con resultados de los estudios genómicos es un recurso valioso que puede complementar el diagnóstico médico y orientar en muchos casos la conducta a seguir. Así se ejemplifica y argumenta en Rehm HL, Berg JS, Brooks LD, Bustamante CD, Evans JP, Landrum MJ, et al. ClinGen — The Clinical Genome Resource. N Engl J Med 2015;372:2235-2242.
Filed under Bases de datos, Bioinformática, Diagnóstico, Genómica, Medicina personalizada, Tratamientos by on . Comment. ![]()
Dos tecnologías de secuenciación, cuyos resultados se obtuvieron en menos de dos horas, permitieron identificar los serotipos implicados en un brote intrahospitalario de Salmonella enterica serovar enteritidis, que afectó a más de 30 pacientes. Puede leer al respecto en Quick J, Ashton P, Calus S, Chatt C, Gossain S, Hawker J, et al. Rapid draft sequencing and real-time nanopore sequencing in a hospital outbreak of Salmonella. Genome Biology 2015;16:114.
Filed under Bioinformática, Diagnóstico, Patógenos by on . Comment. ![]()
 La modelación bioinformática de la expresión diferencial de genes en modelos animales de la enfermedad arterial periférica ha identificado la participación de genes que no se esperaba estuvieran implicados en esta afección, como EphA4, TSPAN7, SLC22A4 y EIF2a. Los hallazgos se comentan en Chu LH, Vijay CG, Annex BH, Bader JS, Popel AS. PADPIN: Protein-Protein Interaction Networks of Angiogenesis, Arteriogenesis, and Inflammation in Peripheral Arterial Disease. Physiological Genomics June 2015; DOI: 10.1152/physiolgenomics.00125.2014.
La modelación bioinformática de la expresión diferencial de genes en modelos animales de la enfermedad arterial periférica ha identificado la participación de genes que no se esperaba estuvieran implicados en esta afección, como EphA4, TSPAN7, SLC22A4 y EIF2a. Los hallazgos se comentan en Chu LH, Vijay CG, Annex BH, Bader JS, Popel AS. PADPIN: Protein-Protein Interaction Networks of Angiogenesis, Arteriogenesis, and Inflammation in Peripheral Arterial Disease. Physiological Genomics June 2015; DOI: 10.1152/physiolgenomics.00125.2014.
Filed under Bioinformática, Diagnóstico by on . Comment. ![]()



