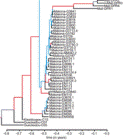
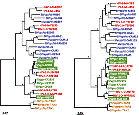
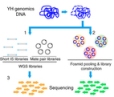
 El análisis por haplotipos del genoma humano ha revelado nuevas secuencias codificadoras, entre ellas al menos seis genes. Los resultados, los más completos hasta el momento, podrían ser útiles en medicina personalizada, de acuerdo con Cao H, Wu H, Luo R, Huang S, Sun Y, Tong X, et al. De novo assembly of a haplotype-resolved human genome. Nature Biotechnology 2015; doi:10.1038/nbt.3200.
El análisis por haplotipos del genoma humano ha revelado nuevas secuencias codificadoras, entre ellas al menos seis genes. Los resultados, los más completos hasta el momento, podrían ser útiles en medicina personalizada, de acuerdo con Cao H, Wu H, Luo R, Huang S, Sun Y, Tong X, et al. De novo assembly of a haplotype-resolved human genome. Nature Biotechnology 2015; doi:10.1038/nbt.3200.
Filed under Bioinformática, Genómica, Medicina personalizada, Proyecto Genoma Humano by on . Comment. ![]()