Web Apollo es un nuevo editor de genomas que puede emplearse con los principales navegadores de internet para hacer anotaciones en línea en tiempo real. Sus características son presentadas en Lee E, Helt G, Reese J, Munoz-Torres MC, Childers C, Buels RM, et al. Web Apollo: a web-based genomic annotation editing platform. Genome Biology 2013;14:R93.
DrGaP se apoya en poderosos métodos estadísticos y algoritmos bioinformáticos para revelar las mutaciones y vías de señalización con un papel clave en los procesos de malignización. El resumen de su descripción puede ser consultado en Hua X, Xu H, Yang Y, Zhu J, Liu P, Lu Y. DrGaP: A Powerful Tool for Identifying Driver Genes and Pathways in Cancer Sequencing Studies. The American Journal of Human Genetics 2013;93(3):439-451.
Filed under Algoritmos, Bioinformática, Cáncer by on . Comment. ![]()
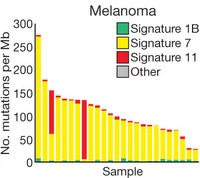 El análisis bioinformático de los datos de secuencias del exoma en más de treinta tipos tumorales comunes ha encontrado al menos veinte firmas genéticas muy frecuentes, en varias combinaciones en la mayoría de las neoplasias malignas. Tales hallazgos son comentados en Alexandrov LB, Nik-Zainal S, Wedge DC, Aparicio SAJR, Behjati S, Biankin AV, et al. Signatures of mutational processes in human cancer. Nature 14 August 2013; doi:10.1038/nature12477.
El análisis bioinformático de los datos de secuencias del exoma en más de treinta tipos tumorales comunes ha encontrado al menos veinte firmas genéticas muy frecuentes, en varias combinaciones en la mayoría de las neoplasias malignas. Tales hallazgos son comentados en Alexandrov LB, Nik-Zainal S, Wedge DC, Aparicio SAJR, Behjati S, Biankin AV, et al. Signatures of mutational processes in human cancer. Nature 14 August 2013; doi:10.1038/nature12477.
Filed under Bioinformática, Cáncer, Diagnóstico, Polimorfismos by on . Comment. ![]()
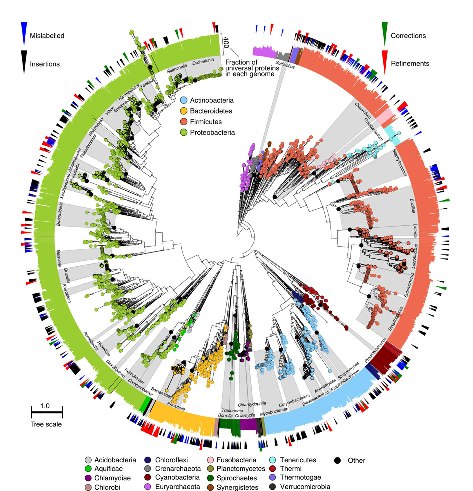 PhyloPhlAn es una aplicación bioinformática para el análisis filogenético y taxonómico que emplea más de 400 proteínas a partir de 3737 genomas microbianos. De código abierto, es presentado en Segata N, Börnigen N, Morgan XC, Huttenhower C. PhyloPhlAn is a new method for improved phylogenetic and taxonomic placement of microbes. Nature Communications 14 August 2013; doi:10.1038/ncomms3304.
PhyloPhlAn es una aplicación bioinformática para el análisis filogenético y taxonómico que emplea más de 400 proteínas a partir de 3737 genomas microbianos. De código abierto, es presentado en Segata N, Börnigen N, Morgan XC, Huttenhower C. PhyloPhlAn is a new method for improved phylogenetic and taxonomic placement of microbes. Nature Communications 14 August 2013; doi:10.1038/ncomms3304.
Filed under Algoritmos, Bioinformática, Filogenética, Patógenos by on . Comment. ![]()
 4273pi es un sistema operativo que ha sido desarrollado a partir de Raspbian Linux con el objetivo de minimizar los requerimientos de administración de sistema para la enseñanza de la bioinformática. Su descripción aparece en Barker D, Ferrier DEK, Holland PWH, Mitchell JBO, Plaisier H, Ritchie MG, et al. 4273pi: Bioinformatics education on low cost ARM hardware. BMC Bioinformatics 2013;14:243.
4273pi es un sistema operativo que ha sido desarrollado a partir de Raspbian Linux con el objetivo de minimizar los requerimientos de administración de sistema para la enseñanza de la bioinformática. Su descripción aparece en Barker D, Ferrier DEK, Holland PWH, Mitchell JBO, Plaisier H, Ritchie MG, et al. 4273pi: Bioinformatics education on low cost ARM hardware. BMC Bioinformatics 2013;14:243.
Filed under Bioinformática, Capacitación by on . Comment. ![]()
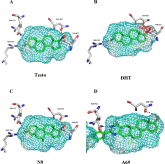 La evaluación virtual de la actividad anabólica de 269 esteroides y la simulación computacional de su interacción con el receptor androgénico humano permitieron identificar las propiedades que favorecen la mejor actividad anabólica, según Alvarez-Ginarte YM, Montero-Cabrera LA, García-de la Vega JM, Bencomo-Martínez A, Pupo A, Agramonte-Delgado A, et al. Integration of ligand and structure-based virtual screening for identification of leading anabolic steroids. J Steroid Biochem Mol Biol. 2013 Jul 18; doi: 10.1016/j.jsbmb.2013.07.004.
La evaluación virtual de la actividad anabólica de 269 esteroides y la simulación computacional de su interacción con el receptor androgénico humano permitieron identificar las propiedades que favorecen la mejor actividad anabólica, según Alvarez-Ginarte YM, Montero-Cabrera LA, García-de la Vega JM, Bencomo-Martínez A, Pupo A, Agramonte-Delgado A, et al. Integration of ligand and structure-based virtual screening for identification of leading anabolic steroids. J Steroid Biochem Mol Biol. 2013 Jul 18; doi: 10.1016/j.jsbmb.2013.07.004.
Filed under Algoritmos, Bioinformática, Biología sintética, Tratamientos by on . Comment. ![]()
 Investigadores de la Universidad Central Marta Abreu de Las Villas participaron en una investigación para la caracterización de los dominios de adenilación de las sintetasas no ribosomales de cianobacterias, disponible en Agüero-Chapin G, Molina-Ruiz R, Maldonado E, de la Riva G, Sánchez-Rodríguez A, Vasconcelos V, et al. Exploring the adenylation domain repertoire of nonribosomal Peptide synthetases using an ensemble of sequence-search methods. PLoS One. 2013 Jul 16;8(7):e65926.
Investigadores de la Universidad Central Marta Abreu de Las Villas participaron en una investigación para la caracterización de los dominios de adenilación de las sintetasas no ribosomales de cianobacterias, disponible en Agüero-Chapin G, Molina-Ruiz R, Maldonado E, de la Riva G, Sánchez-Rodríguez A, Vasconcelos V, et al. Exploring the adenylation domain repertoire of nonribosomal Peptide synthetases using an ensemble of sequence-search methods. PLoS One. 2013 Jul 16;8(7):e65926.
Filed under Algoritmos, Bioinformática by on . Comment. ![]()
![]() La secuenciación de tres docenas de cepas de virus influenza A H7N9, tomadas de aves y mercados avícolas en China, ha arrojado luz sobre los mínimos cambios que se requieren para una mayor virulencia y su transmisión a humanos. El reporte aparece en Zhang Q, Shi J, Deng G, Guo J, Zeng X, He X, et al. H7N9 Influenza Viruses Are Transmissible in Ferrets by Respiratory Droplet. Science 26 July 2013;341(6144):410-414.
La secuenciación de tres docenas de cepas de virus influenza A H7N9, tomadas de aves y mercados avícolas en China, ha arrojado luz sobre los mínimos cambios que se requieren para una mayor virulencia y su transmisión a humanos. El reporte aparece en Zhang Q, Shi J, Deng G, Guo J, Zeng X, He X, et al. H7N9 Influenza Viruses Are Transmissible in Ferrets by Respiratory Droplet. Science 26 July 2013;341(6144):410-414.
Filed under Bioinformática, Diagnóstico, Filogenética, Patógenos by on . Comment. ![]()
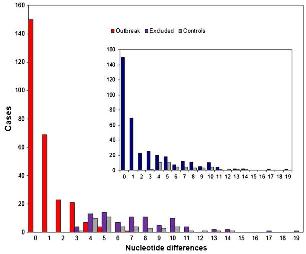 El análisis filogenético de las secuencias de dos regiones del genoma del virus de la hepatitis C, obtenidas de 322 pacientes relacionados, permitió excluir del brote a 47 enfermos, al tiempo que confirmó como responsable de la infección a un anestesista sometido a juicio por esa razón. Puede leer el reporte completo en González-Candelas F, Bracho MA, Wróbel B, Moya A. Molecular evolution in court: analysis of a large hepatitis C virus outbreak from an evolving source. BMC Biology 2013;11:76.
El análisis filogenético de las secuencias de dos regiones del genoma del virus de la hepatitis C, obtenidas de 322 pacientes relacionados, permitió excluir del brote a 47 enfermos, al tiempo que confirmó como responsable de la infección a un anestesista sometido a juicio por esa razón. Puede leer el reporte completo en González-Candelas F, Bracho MA, Wróbel B, Moya A. Molecular evolution in court: analysis of a large hepatitis C virus outbreak from an evolving source. BMC Biology 2013;11:76.
Filed under Bioinformática, Diagnóstico, Filogenética, Patógenos by on . Comment. ![]()
 Java BioWareHouse (JBioWH) es una herramienta creada por investigadores cubanos y de otros países, que permite recuperar datos de bases como NCBI, KEGG, así como analizar resultados de experimentos de alto flujo, entre otras aplicaciones. JBioWH es descrita en Vera R, Perez-Rivero Y, Perez S, Ligeti B, Kertész-Farkas A, Pongor S. JBioWH: an open-source Java framework for bioinformatics data integration. Database 2013:bat051, doi: 10.1093/database/bat051.
Java BioWareHouse (JBioWH) es una herramienta creada por investigadores cubanos y de otros países, que permite recuperar datos de bases como NCBI, KEGG, así como analizar resultados de experimentos de alto flujo, entre otras aplicaciones. JBioWH es descrita en Vera R, Perez-Rivero Y, Perez S, Ligeti B, Kertész-Farkas A, Pongor S. JBioWH: an open-source Java framework for bioinformatics data integration. Database 2013:bat051, doi: 10.1093/database/bat051.
Filed under Algoritmos, Bioinformática, Genómica by on . Comment. ![]()



