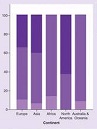
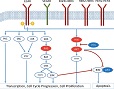
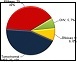
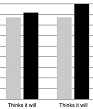
 Una encuesta impresa y en línea ha revelado que los pediatras norteamericanos y japoneses tienen un bajo conocimiento sobre la farmacogenética, aunque creen que es importante su implementación y tienen disposición para recibir formación al respecto. Así aparece en Rahawi S, Naik H, Blake KV, Obeng AO, Wasserman RM, Seki Y, et al. Knowledge and attitudes on pharmacogenetics among pediatricians. Journal of Human Genetics 2020.
Una encuesta impresa y en línea ha revelado que los pediatras norteamericanos y japoneses tienen un bajo conocimiento sobre la farmacogenética, aunque creen que es importante su implementación y tienen disposición para recibir formación al respecto. Así aparece en Rahawi S, Naik H, Blake KV, Obeng AO, Wasserman RM, Seki Y, et al. Knowledge and attitudes on pharmacogenetics among pediatricians. Journal of Human Genetics 2020.
Filed under Capacitación, Farmacogenómica by on . Comment. ![]()