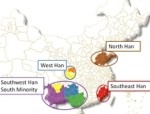
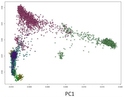
La comparación genómica entre Mycobacterium leprae y M. lepromatosis condujo a determinar que ambos gérmenes derivan de un ancestro común, del que divergieron hace cerca de 14 millones de años, tras lo cual han continuado perdiendo genes. El trabajo original está disponible en Singh P, Benjak A, Schuenemann VJ, Herbig A, Avanzi C, Busso P, et al. Insight into the evolution and origin of leprosy bacilli from the genome sequence of Mycobacterium lepromatosis. PNAS March 23, 2015; doi: 10.1073/pnas.1421504112.
La comparación genómica entre Mycobacterium leprae y M. lepromatosis condujo a determinar que ambos gérmenes derivan de un ancestro común, del que divergieron hace cerca de 14 millones de años, tras lo cual han continuado perdiendo genes. El trabajo original está disponible en Singh P, Benjak A, Schuenemann VJ, Herbig A, Avanzi C, Busso P, et al. Insight into the evolution and origin of leprosy bacilli from the genome sequence of Mycobacterium lepromatosis. PNAS March 23, 2015; doi: 10.1073/pnas.1421504112.
Filed under Bioinformática, Filogenética, Patógenos by on . Comment. ![]()