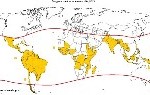
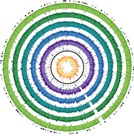
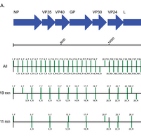
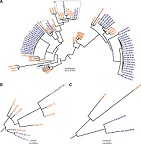
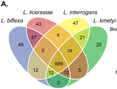
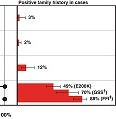
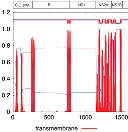
 La comparación de las secuencias reportadas para cepas de virus antes y después de la actual epidemia ha identificado algunas diferencias para el actual brote. Lea al respecto en Zhu Z, Chan JFW, Tee KM, Choi GKY, Lau SKP, Woo PCY, et al. Comparative genomic analysis of pre-epidemic and epidemic Zika virus strains for virological factors potentially associated with the rapidly expanding epidemic. Emerging Microbes & Infections 2016;5:e22.
La comparación de las secuencias reportadas para cepas de virus antes y después de la actual epidemia ha identificado algunas diferencias para el actual brote. Lea al respecto en Zhu Z, Chan JFW, Tee KM, Choi GKY, Lau SKP, Woo PCY, et al. Comparative genomic analysis of pre-epidemic and epidemic Zika virus strains for virological factors potentially associated with the rapidly expanding epidemic. Emerging Microbes & Infections 2016;5:e22.
Filed under Bioinformática, Filogenética, Patógenos by on . Comment. ![]()