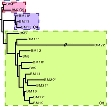
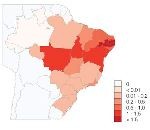
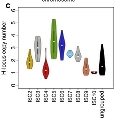
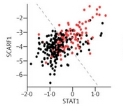
 Para avivar el debate ético sobre la introducción de modificaciones en embriones humanos a través de la edición del genoma, se ha reportado la introducción en cigotos de una secuencia específica para limitar la infección por vih. Puede consultarlo en Kang X, He W, Huang Y, Yu Q, Chen Y, Gao X, et al. Introducing precise genetic modifications into human 3PN embryos by CRISPR/Cas-mediated genome editing. Journal of Assisted Reproduction and Genetics May 2016;33(5):581–588.
Para avivar el debate ético sobre la introducción de modificaciones en embriones humanos a través de la edición del genoma, se ha reportado la introducción en cigotos de una secuencia específica para limitar la infección por vih. Puede consultarlo en Kang X, He W, Huang Y, Yu Q, Chen Y, Gao X, et al. Introducing precise genetic modifications into human 3PN embryos by CRISPR/Cas-mediated genome editing. Journal of Assisted Reproduction and Genetics May 2016;33(5):581–588.
Filed under Edición del genoma, Medicina personalizada, Patógenos by on . Comment. ![]()