La piel poco pigmentada ya abundaba entre los individuos europeos de la Edad de Bronce, pero no la tolerancia a la lactosa, de acuerdo con la secuenciación genómica de 101 sujetos de esa época, que se publica en Allentoft ME, Sikora M, Sjögren KG, Rasmussen S, Rasmussen M, Stenderup J, et al. Population genomics of Bronze Age Eurasia. Nature 11 June 2015;522:167–172.
Un estudio de pobladores de Islandia (Gudbjartsson DF, Helgason H, Gudjonsson SA, Zink F, Oddson A, Gylfason A, et al. Large-scale whole-genome sequencing of the Icelandic population. Nature Genetics 2015; doi:10.1038/ng.3247) ha encontrado variantes de susceptibilidad para enfermedades cardiacas, hepáticas y tiroideas. La contribución de Europa y África a la mezcla que se produjo en las poblaciones americanas es abordada en Montinaro F, Busby GBJ, Pascali VL, Myers S, Hellenthal G, Capelli C. Unravelling the hidden ancestry of American admixed populations. Nature Communications 2015;6:6596, doi:10.1038/ncomms7596.
Filed under Bioinformática, Diagnóstico, Filogenética, Genómica by on . Comment. ![]()
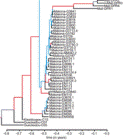 La tasa de mutaciones en muestras del virus ébola aislado en Mali no aporta evidencias para una rápida evolución genética en el actual brote de África. Las implicaciones para la salud pública son comentadas en Hoenen T, Safronetz D, Groseth A, Wollenberg KR, Koita OA, Diarra B, et al. Mutation rate and genotype variation of Ebola virus from Mali case sequences. Science 2015; DOI: 10.1126/science.aaa5646.
La tasa de mutaciones en muestras del virus ébola aislado en Mali no aporta evidencias para una rápida evolución genética en el actual brote de África. Las implicaciones para la salud pública son comentadas en Hoenen T, Safronetz D, Groseth A, Wollenberg KR, Koita OA, Diarra B, et al. Mutation rate and genotype variation of Ebola virus from Mali case sequences. Science 2015; DOI: 10.1126/science.aaa5646.
Filed under Bioinformática, Filogenética, Patógenos, Polimorfismos by on . Comment. ![]()
 La comparación genómica entre Mycobacterium leprae y M. lepromatosis condujo a determinar que ambos gérmenes derivan de un ancestro común, del que divergieron hace cerca de 14 millones de años, tras lo cual han continuado perdiendo genes. El trabajo original está disponible en Singh P, Benjak A, Schuenemann VJ, Herbig A, Avanzi C, Busso P, et al. Insight into the evolution and origin of leprosy bacilli from the genome sequence of Mycobacterium lepromatosis. PNAS March 23, 2015; doi: 10.1073/pnas.1421504112.
La comparación genómica entre Mycobacterium leprae y M. lepromatosis condujo a determinar que ambos gérmenes derivan de un ancestro común, del que divergieron hace cerca de 14 millones de años, tras lo cual han continuado perdiendo genes. El trabajo original está disponible en Singh P, Benjak A, Schuenemann VJ, Herbig A, Avanzi C, Busso P, et al. Insight into the evolution and origin of leprosy bacilli from the genome sequence of Mycobacterium lepromatosis. PNAS March 23, 2015; doi: 10.1073/pnas.1421504112.
Filed under Bioinformática, Filogenética, Patógenos by on . Comment. ![]()
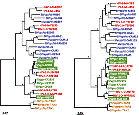 El análisis filogenético de cepas del virus de inmunodeficiencia de los simios (SIV) en muestras obtenidas de gorilas de Camerún ha encontrado secuencias ancestrales de las que pudo haber derivado el vih-1 del grupo O. El tema es discutido en D’arc M, Ayouba A, Esteban A, Learn GH, Boué V, Liegeois F, et al. Origin of the HIV-1 group O epidemic in western lowland gorillas. PNAS 2015;112(11):E1343–E1352.
El análisis filogenético de cepas del virus de inmunodeficiencia de los simios (SIV) en muestras obtenidas de gorilas de Camerún ha encontrado secuencias ancestrales de las que pudo haber derivado el vih-1 del grupo O. El tema es discutido en D’arc M, Ayouba A, Esteban A, Learn GH, Boué V, Liegeois F, et al. Origin of the HIV-1 group O epidemic in western lowland gorillas. PNAS 2015;112(11):E1343–E1352.
Filed under Bioinformática, Filogenética by on . Comment. ![]()
Por medio de una estrategia bioinformática basada en el análisis filogenético de secuencias de más de un centenar de otras especies eucariotas, se proponen funciones para cientos de genes humanos hasta ahora poco caracterizados. Lea el reporte en Dey G, Jaimovich A, Collins SR, Seki A, Meyer T. Systematic Discovery of Human Gene Function and Principles of Modular Organization through Phylogenetic Profiling. Cell Reports 17 February 2015;10(6):993–1006.
Filed under Bioinformática, Filogenética by on . Comment. ![]()
![]() La Universidad de California Santa Cruz ha incluido en su colección de recursos genómicos las secuencias virales de brotes anteriores y de la actual epidemia de ébola, junto a herramientas bioinformáticas para su estudio. Pueden ser consultados en el UCSC Ebola Genome Portal.
La Universidad de California Santa Cruz ha incluido en su colección de recursos genómicos las secuencias virales de brotes anteriores y de la actual epidemia de ébola, junto a herramientas bioinformáticas para su estudio. Pueden ser consultados en el UCSC Ebola Genome Portal.
Filed under Algoritmos, Bases de datos, Bioinformática, Filogenética, Patógenos by on . Comment. ![]()
 La secuenciación del genoma completo fue usada para establecer las relaciones entre 114 cepas de Acinetobacter baumannii en un brote hospitalario del germen. La identificación de las fuentes de contaminación es uno de los resultados, sobre los que puede leer en Halachev MR, Chan JZM, Constantinidou CI, Cumley N, Bradley C, Smith-Banks M, et al. Genomic epidemiology of a protracted hospital outbreak caused by multidrug-resistant Acinetobacter baumannii in Birmingham, England. Genome Medicine 2014;6:70.
La secuenciación del genoma completo fue usada para establecer las relaciones entre 114 cepas de Acinetobacter baumannii en un brote hospitalario del germen. La identificación de las fuentes de contaminación es uno de los resultados, sobre los que puede leer en Halachev MR, Chan JZM, Constantinidou CI, Cumley N, Bradley C, Smith-Banks M, et al. Genomic epidemiology of a protracted hospital outbreak caused by multidrug-resistant Acinetobacter baumannii in Birmingham, England. Genome Medicine 2014;6:70.
Filed under Diagnóstico, Filogenética, Patógenos by on . Comment. ![]()
 312 cepas de Escherichia coli obtenidas de la sangre o la orina fueron secuenciadas para revelar las diferencias en la resistencia a antibióticos, los sitios de infección y los factores de virulencia. Los hallazgos son descritos en Salipante SJ, Roach DJ, Kitzman JO, Snyder, Stackhouse B, Butler-Wu SM, et al. Large-scale genomic sequencing of extraintestinal pathogenic Escherichia coli strains. Genome Res. November 4, 2014; doi: 10.1101/gr.180190.114.
312 cepas de Escherichia coli obtenidas de la sangre o la orina fueron secuenciadas para revelar las diferencias en la resistencia a antibióticos, los sitios de infección y los factores de virulencia. Los hallazgos son descritos en Salipante SJ, Roach DJ, Kitzman JO, Snyder, Stackhouse B, Butler-Wu SM, et al. Large-scale genomic sequencing of extraintestinal pathogenic Escherichia coli strains. Genome Res. November 4, 2014; doi: 10.1101/gr.180190.114.
Filed under Filogenética, Patógenos by on . Comment. ![]()
 Las muestras de 78 enfermos infectados en la actual epidemia de ébola permitieron aislar 99 secuencias virales y reconstruir la historia del brote: derivada de una cepa de África central de 2004, cruzó de Guinea a Sierra Leona en mayo de 2014 con una sostenida transmisión entre humanos sin evidencia de otras fuentes zoonóticas adicionales. Las implicaciones de las mutaciones para el diagnóstico, las vacunas y los tratamientos son comentados en Gire SK, Goba A, Andersen, Sealfon RSG, Park DJ, Kanneh L, Jalloh S, et al. Genomic surveillance elucidates Ebola virus origin and transmission during the 2014 outbreak. Science 12 September 2014;345(6202):1369-1372.
Las muestras de 78 enfermos infectados en la actual epidemia de ébola permitieron aislar 99 secuencias virales y reconstruir la historia del brote: derivada de una cepa de África central de 2004, cruzó de Guinea a Sierra Leona en mayo de 2014 con una sostenida transmisión entre humanos sin evidencia de otras fuentes zoonóticas adicionales. Las implicaciones de las mutaciones para el diagnóstico, las vacunas y los tratamientos son comentados en Gire SK, Goba A, Andersen, Sealfon RSG, Park DJ, Kanneh L, Jalloh S, et al. Genomic surveillance elucidates Ebola virus origin and transmission during the 2014 outbreak. Science 12 September 2014;345(6202):1369-1372.
Filed under Diagnóstico, Filogenética, Otros Proyectos Genoma, Patógenos by on . Comment. ![]()



