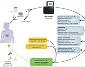
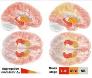
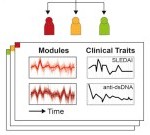
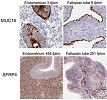
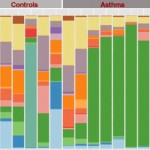
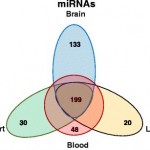
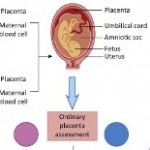
Los análisis de expresión de 415 genes permitió definir un índice de severidad para la esclerosis sistémica y distinguir precozmente los enfermos respondedores de los no respondedores. Puede ver los resultados en Lofgren S, Hinchcliff M, Carns M, Wood T, Aren K, Arroyo E, et al. Integrated, multicohort analysis of systemic sclerosis identifies robust transcriptional signature of disease severity. JCI Insight. 2016;1(21):e89073.
Los análisis de expresión de 415 genes permitió definir un índice de severidad para la esclerosis sistémica y distinguir precozmente los enfermos respondedores de los no respondedores. Puede ver los resultados en Lofgren S, Hinchcliff M, Carns M, Wood T, Aren K, Arroyo E, et al. Integrated, multicohort analysis of systemic sclerosis identifies robust transcriptional signature of disease severity. JCI Insight. 2016;1(21):e89073.
Filed under Diagnóstico, Medicina personalizada, Transcriptómica by on . Comment. ![]()