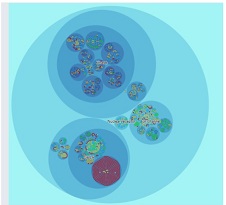
Un nuevo grupo de virus gigantes, denominado como Klosneuviruses, ha sido descubierto en datos metagenómicos. No parece que evolucionaran a partir de un ancestro celular sino de otros virus más pequeños. Puede leer el original en Schulz F, Yutin N, Ivanova NN, Ortega DR, Lee TK, Vierheilig J, et al. Giant viruses with an expanded complement of translation system components. Science 2017;356(6333):82-85.
Un nuevo grupo de virus gigantes, denominado como Klosneuviruses, ha sido descubierto en datos metagenómicos. No parece que evolucionaran a partir de un ancestro celular sino de otros virus más pequeños. Puede leer el original en Schulz F, Yutin N, Ivanova NN, Ortega DR, Lee TK, Vierheilig J, et al. Giant viruses with an expanded complement of translation system components. Science 2017;356(6333):82-85.
Filed under Bioinformática, Filogenética, Patógenos by on . Comment. ![]()