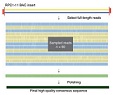
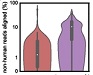
El catálogo GWAS, mantenido por el Instituto Europeo de Bioinformática, cumple el 28 de abril su décimo aniversario. Contiene casi 5000 trabajos publicados y cerca de 70000 asociaciones entre polimorfismos genéticos y enfermedades humanas o rasgos no patológicos. Puede consultar este excelente recurso en su sitio web.
El catálogo GWAS, mantenido por el Instituto Europeo de Bioinformática, cumple el 28 de abril su décimo aniversario. Contiene casi 5000 trabajos publicados y cerca de 70000 asociaciones entre polimorfismos genéticos y enfermedades humanas o rasgos no patológicos. Puede consultar este excelente recurso en su sitio web.
Filed under Bases de datos, Bioinformática, Diagnóstico, Polimorfismos by on . Comment. ![]()