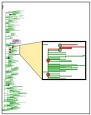
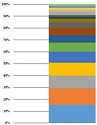
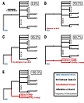
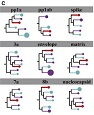
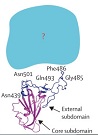
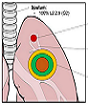
 Muestras pulmonares obtenidas por cirugía de pacientes tuberculosos tuvieron un 40 % de infecciones por al menos dos cepas no relacionadas, a partir de la secuenciación genómica, lo que puede pasar inadvertido en muestras de esputo. Las implicaciones clínicas son discutidas en Moreno-Molina M, Shubladze N, Khurtsilava I, Avaliani Z, Bablishvili N, Torres-Puente M, et al. Genomic analyses of Mycobacterium tuberculosis from human lung resections reveal a high frequency of polyclonal infections. Nature Communications. 2021;12:2716.
Muestras pulmonares obtenidas por cirugía de pacientes tuberculosos tuvieron un 40 % de infecciones por al menos dos cepas no relacionadas, a partir de la secuenciación genómica, lo que puede pasar inadvertido en muestras de esputo. Las implicaciones clínicas son discutidas en Moreno-Molina M, Shubladze N, Khurtsilava I, Avaliani Z, Bablishvili N, Torres-Puente M, et al. Genomic analyses of Mycobacterium tuberculosis from human lung resections reveal a high frequency of polyclonal infections. Nature Communications. 2021;12:2716.
Filed under Diagnóstico, Filogenética, Patógenos by on . Comment. ![]()