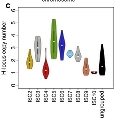
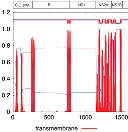
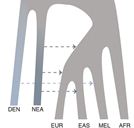
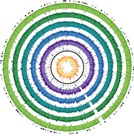
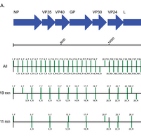
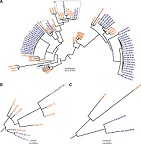
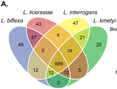
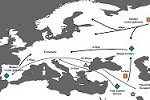
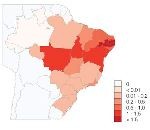
 Los análisis filogenéticos sugieren una única introducción del virus del zika en las Américas, así como sus vías de diseminación a partir de Brasil. Estos y otros hallazgos son descritos en Rodrigues Faria N, do Socorro R, Kraemer MUG, Souza R, Sequetin Cunha M, Hill SC, et al. Zika virus in the Americas: Early epidemiological and genetic findings. Science 24 Mar 2016; DOI: 10.1126/science.aaf5036.
Los análisis filogenéticos sugieren una única introducción del virus del zika en las Américas, así como sus vías de diseminación a partir de Brasil. Estos y otros hallazgos son descritos en Rodrigues Faria N, do Socorro R, Kraemer MUG, Souza R, Sequetin Cunha M, Hill SC, et al. Zika virus in the Americas: Early epidemiological and genetic findings. Science 24 Mar 2016; DOI: 10.1126/science.aaf5036.
Filed under Bioinformática, Filogenética, Patógenos by on . Comment. ![]()