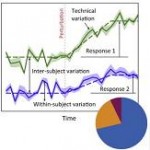
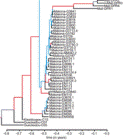
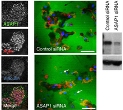
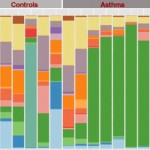
 Técnicas de secuenciación metagenómica de ARN han identificado poblaciones bacterianas más abundantes en la cavidad nasal de niños asmáticos, en relación con controles sanos. Moraxella catarrhalis es la más representada, de acuerdo con Castro-Nallar E, Shen Y, Freishtat RJ, Pérez-Losada M, Manimaran S, Liu G, et al. Integrating microbial and host transcriptomics to characterize asthma-associated microbial communities. BMC Medical Genomics 2015;8:50.
Técnicas de secuenciación metagenómica de ARN han identificado poblaciones bacterianas más abundantes en la cavidad nasal de niños asmáticos, en relación con controles sanos. Moraxella catarrhalis es la más representada, de acuerdo con Castro-Nallar E, Shen Y, Freishtat RJ, Pérez-Losada M, Manimaran S, Liu G, et al. Integrating microbial and host transcriptomics to characterize asthma-associated microbial communities. BMC Medical Genomics 2015;8:50.
Filed under Diagnóstico, Microbioma, Patógenos, Transcriptómica by on . Comment. ![]()