Por medio de una estrategia bioinformática basada en el análisis filogenético de secuencias de más de un centenar de otras especies eucariotas, se proponen funciones para cientos de genes humanos hasta ahora poco caracterizados. Lea el reporte en Dey G, Jaimovich A, Collins SR, Seki A, Meyer T. Systematic Discovery of Human Gene Function and Principles of Modular Organization through Phylogenetic Profiling. Cell Reports 17 February 2015;10(6):993–1006.
 En el 2015 culminará la iniciativa The Cancer Genome Atlas, tras nueve años dedicados a la búsqueda de una mejor comprensión de la biología tumoral y su traducción al diagnóstico y las terapias. Al respecto, y sus nuevos objetivos, se ha publicado el editorial The future of cancer genomics. Nature Medicine 2015;21:99.
En el 2015 culminará la iniciativa The Cancer Genome Atlas, tras nueve años dedicados a la búsqueda de una mejor comprensión de la biología tumoral y su traducción al diagnóstico y las terapias. Al respecto, y sus nuevos objetivos, se ha publicado el editorial The future of cancer genomics. Nature Medicine 2015;21:99.
Filed under Bioinformática, Cáncer, Diagnóstico, Genómica by on . Comment. ![]()
La combinación de técnicas genómicas y bioinformáticas en sujetos con diferente ascendencia, en este caso con ancestros europeos o asiáticos, reveló dos nuevas variantes (rs10816625 y rs13294895) y sus efectos en la génesis del cáncer de mama. El resumen del hallazgo está disponible en Orr N, Dudbridge F, Dryden N, Maguire S, Novo D, Perrakis E, et al. Fine-mapping identifies two additional breast cancer susceptibility loci at 9q31.2. Hum. Mol. Genet. 2015; doi: 10.1093/hmg/ddv035.
Filed under Bioinformática, Cáncer, Diagnóstico, Genómica, Polimorfismos by on . Comment. ![]()
![]() La Universidad de California Santa Cruz ha incluido en su colección de recursos genómicos las secuencias virales de brotes anteriores y de la actual epidemia de ébola, junto a herramientas bioinformáticas para su estudio. Pueden ser consultados en el UCSC Ebola Genome Portal.
La Universidad de California Santa Cruz ha incluido en su colección de recursos genómicos las secuencias virales de brotes anteriores y de la actual epidemia de ébola, junto a herramientas bioinformáticas para su estudio. Pueden ser consultados en el UCSC Ebola Genome Portal.
Filed under Algoritmos, Bases de datos, Bioinformática, Filogenética, Patógenos by on . Comment. ![]()
 Numerosos ejemplos de bases de datos con información general sobre el cáncer, algunos recursos relacionados con tipos tumorales específicos y las herramientas bioinformáticas para extraer información de ellas, son incluidos en Pavlopoulou A, Spandidos DA, Michalopoulos I. Human cancer databases. Oncol Rep. 2014 Oct 31. doi: 10.3892/or.2014.3579.
Numerosos ejemplos de bases de datos con información general sobre el cáncer, algunos recursos relacionados con tipos tumorales específicos y las herramientas bioinformáticas para extraer información de ellas, son incluidos en Pavlopoulou A, Spandidos DA, Michalopoulos I. Human cancer databases. Oncol Rep. 2014 Oct 31. doi: 10.3892/or.2014.3579.
Filed under Bases de datos, Bioinformática, Cáncer by on . Comment. ![]()
 La inclusión de un parámetro adicional en un nuevo modelo que incrementa la capacidad para definir la asociación en los estudios de genoma completo, selecciona la mejor combinación entre los algoritmos de parentesco y agrupación. La nueva propuesta es descrita en Li M, Liu X, Bradbury P, Yu J, Zhang Y-M, Todhunter RJ, et al. Enrichment of statistical power for genome-wide association studies. BMC Biology 2014;12:73.
La inclusión de un parámetro adicional en un nuevo modelo que incrementa la capacidad para definir la asociación en los estudios de genoma completo, selecciona la mejor combinación entre los algoritmos de parentesco y agrupación. La nueva propuesta es descrita en Li M, Liu X, Bradbury P, Yu J, Zhang Y-M, Todhunter RJ, et al. Enrichment of statistical power for genome-wide association studies. BMC Biology 2014;12:73.
Filed under Algoritmos, Bioinformática, Diagnóstico, Genómica by on . Comment. ![]()
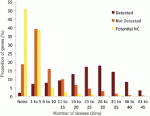 El empleo de técnicas proteómicas ha llevado a cuestionar si muchas secuencias génicas son realmente codificadoras y, por la misma razón, a estimar que el genoma humano contiene un número menor de genes, calculado ahora en alrededor de 19000. Así se presenta en Ezkurdia I, Juan D, Rodriguez JM, Frankish A, Diekhans M, Harrow J, et al. Multiple evidence strands suggest that there may be as few as 19 000 human protein-coding genes. Hum Mol Genet. 2014 Jun 16; doi: 10.1093/hmg/ddu309.
El empleo de técnicas proteómicas ha llevado a cuestionar si muchas secuencias génicas son realmente codificadoras y, por la misma razón, a estimar que el genoma humano contiene un número menor de genes, calculado ahora en alrededor de 19000. Así se presenta en Ezkurdia I, Juan D, Rodriguez JM, Frankish A, Diekhans M, Harrow J, et al. Multiple evidence strands suggest that there may be as few as 19 000 human protein-coding genes. Hum Mol Genet. 2014 Jun 16; doi: 10.1093/hmg/ddu309.
Filed under Bioinformática, Genómica, Proteómica, Proyecto Genoma Humano by on . Comment. ![]()
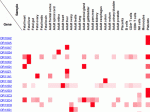 Los dos estudios recientemente publicados sobre la caracterización del proteoma humano deben ser tomados con cautela, pues se han encontrado inconsistencias a partir del análisis de la familia de los receptores olfatorios. Tal es la opinión central de Ezkurdia I, Vázquez J, Valencia A, Tress M. Analyzing the First Drafts of the Human Proteome. J Proteome Res 2014;13(8):3854–3855.
Los dos estudios recientemente publicados sobre la caracterización del proteoma humano deben ser tomados con cautela, pues se han encontrado inconsistencias a partir del análisis de la familia de los receptores olfatorios. Tal es la opinión central de Ezkurdia I, Vázquez J, Valencia A, Tress M. Analyzing the First Drafts of the Human Proteome. J Proteome Res 2014;13(8):3854–3855.
Filed under Bioinformática, Proteómica by on . Comment. ![]()
El análisis del proteoma de Trypanosoma cruzi, agente causal de la enfermedad de Chagas, permitió seleccionar un grupo de secuencias que son candidatas en el diseño de una vacuna contra el protozoario. El procedimiento es descrito en Teh-Poot C, Tzec-Arjona E, Martinez-Vega P, Ramirez-Sierra MJ, Rosado-Vallado M, Dumonteil E. From genome to a vaccine against Trypanosoma cruzi by immunoinformatics. J Infect Dis. 2014 Jul 28; doi: 10.1093/infdis/jiu418.
Filed under Bioinformática, Inmunoinformática by on . Comment. ![]()
 The Personal Genome Browser (PGB) permite el análisis de los genomas individuales y destaca las variaciones potenciales en todo el genoma, en un cromosoma o en una citobanda. Sus posibilidades son explicadas en Juan L, Teng M, Zang T, Hao Y, Wang Z, Yan C, et al. The personal genome browser: visualizing functions of genetic variants. Nucl. Acids Res. 2014; doi: 10.1093/nar/gku361.
The Personal Genome Browser (PGB) permite el análisis de los genomas individuales y destaca las variaciones potenciales en todo el genoma, en un cromosoma o en una citobanda. Sus posibilidades son explicadas en Juan L, Teng M, Zang T, Hao Y, Wang Z, Yan C, et al. The personal genome browser: visualizing functions of genetic variants. Nucl. Acids Res. 2014; doi: 10.1093/nar/gku361.
Filed under Bioinformática, Diagnóstico, Genómica, Polimorfismos, Proyecto Genoma Humano by on . Comment. ![]()



