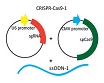
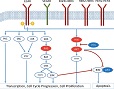
Un algoritmo bioinformático que permite diseñar fragmentos variables funcionales y estables de anticuerpos, con afinidades nanomolares para sus ligandos, podría aplicarse al diseño de otras estructuras, como enzimas, de acuerdo con la opinión de los autores del estudio Baran D, Pszolla MG, Lapidoth GD, Norn C, Dym O, Unger T, et al. Principles for computational design of binding antibodies. PNAS 2017;114(41):10900–10905.
Un algoritmo bioinformático que permite diseñar fragmentos variables funcionales y estables de anticuerpos, con afinidades nanomolares para sus ligandos, podría aplicarse al diseño de otras estructuras, como enzimas, de acuerdo con la opinión de los autores del estudio Baran D, Pszolla MG, Lapidoth GD, Norn C, Dym O, Unger T, et al. Principles for computational design of binding antibodies. PNAS 2017;114(41):10900–10905.
Filed under Algoritmos, Bioinformática, Inmunoinformática, Tratamientos by on . Comment. ![]()