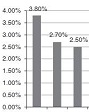
La detección de los genes BRCA a nivel poblacional puede prevenir decenas de miles de casos de cáncer de mama y ovario, a un costo económico que pueden asumir países de ingresos medios y altos, de acuerdo con Manchanda R, Sun L, Patel S, Evans O, Wilschut J, De Freitas Lopes AC, et al. Economic Evaluation of Population-Based BRCA1/BRCA2 Mutation Testing across Multiple Countries and Health Systems. Cancers 2020;12(7):1929.
La detección de los genes BRCA a nivel poblacional puede prevenir decenas de miles de casos de cáncer de mama y ovario, a un costo económico que pueden asumir países de ingresos medios y altos, de acuerdo con Manchanda R, Sun L, Patel S, Evans O, Wilschut J, De Freitas Lopes AC, et al. Economic Evaluation of Population-Based BRCA1/BRCA2 Mutation Testing across Multiple Countries and Health Systems. Cancers 2020;12(7):1929.
Filed under Cáncer, Diagnóstico, Polimorfismos by on . Comment. ![]()